
新技术突破质谱分析瓶颈,为帕金森病等神经退行性疾病研究开辟新路径
蛋白质是生命活动的执行者,其功能的发挥依赖于精确的结构变化和动态调控。然而,长期以来,科学家只能在体外纯化环境中观察这些变化,无法直接"看到"蛋白质在真实细胞环境中的行为。近日,南开大学化学学院李功玉教授团队在国际顶级化学期刊《德国应用化学》发表研究成果,报道了一种名为RapiBOX-IM-MS的创新技术平台,首次实现了在细胞裂解液乃至活细胞中,以亚秒级时间分辨率追踪蛋白质结构动态变化的能力。
困扰领域多年的"基质效应"难题
质谱技术凭借其高灵敏度和高分辨率,已成为研究蛋白质结构与相互作用的重要工具。然而,当研究对象从纯化蛋白转向真实生物样品时,一道难以逾越的障碍便横亘在研究者面前:细胞内大量存在的盐离子、代谢物和脂质会产生严重的信号干扰,即所谓的"基质效应",使得蛋白质信号被完全淹没。
更具挑战性的是,如果想要观察蛋白质对刺激的动态响应,就必须通过快速混合或温度变化来触发反应。但在复杂的细胞基质中,这些操作反而会加剧信号抑制,使得那些转瞬即逝的中间态更加难以捕获。这形成了一个分析化学中的悖论:启动动态过程所必需的方法,恰恰与复杂样品的直接分析互不兼容。
双重机制破解分析悖论
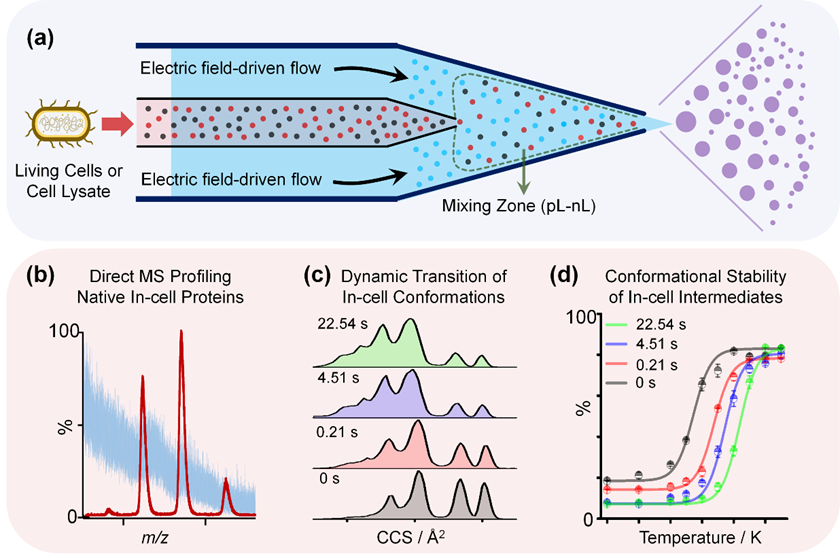
李功玉团队开发的RapiBOX平台巧妙地解决了这一难题。该装置在纳米电喷雾发射端集成了两项关键技术:快速在线缓冲液交换和脉冲电场电泳分离。当样品溶液到达发射端时,系统首先将其与挥发性缓冲液快速混合,随后利用脉冲电场将蛋白质与残留的小分子干扰物分离——整个过程在离子化的瞬间完成。
这种双重机制带来了显著的分析性能提升。研究团队使用含25毫摩尔氯化钠的高盐溶液测试血清白蛋白和β-乳球蛋白,传统纳米电喷雾方法完全无法检测到信号,而RapiBOX不仅成功获得了高质量谱图,信号强度更是提升了一个数量级以上。在细菌裂解液的直接分析中,该平台成功鉴定出48种内源性蛋白质,展现了其在复杂生物基质中进行结构蛋白质组学研究的潜力。
捕获血红蛋白折叠的不对称中间体
该平台最引人注目的能力在于其亚秒级的时间分辨率。通过精确调控混合区域的几何结构,研究团队可以在0.21秒至数十秒的时间窗口内获取蛋白质结构变化的"快照"。
以血红蛋白为模型系统,研究者首次观察到了折叠与去折叠过程中的路径不对称性。在酸诱导的去折叠过程中,血红蛋白四聚体直接解离为二聚体再到单体,未检测到任何同源二聚体中间态。然而在重新折叠过程中,α链和β链的同源二聚体作为短暂存在的中间体清晰可见——这些物种仅在最初几秒内出现,随后迅速转化为稳定的αβ异源二聚体。这一发现揭示了蛋白质折叠过程远比想象中复杂,不同方向的路径可能经历完全不同的能量景观。
揭示α-突触核蛋白的反常行为与致病机制
α-突触核蛋白是帕金森病的核心致病蛋白,其聚集是疾病发生的主要驱动因素。与结构规整的球状蛋白不同,这种内在无序蛋白在酸性条件下表现出截然相反的行为:不是展开,而是迅速压缩。
研究团队在细胞裂解液中直接追踪了这一过程。离子淌度数据清晰显示,随着酸暴露时间的延长,蛋白质群体迅速向紧凑构象富集。这种反常行为源于酸性残基的质子化减少了静电排斥,使得多肽链能够塌缩成更致密的结构。
更具启发性的发现来自于酸和热双重压力的协同效应实验。研究团队开发了温控RapiBOX系统,发现预先经历短暂酸处理的α-突触核蛋白对随后的热变性表现出更强的抵抗能力。这意味着酸诱导的紧凑构象实际上是动力学上被"锁定"的状态,需要更高的热能才能展开。
这一发现具有重要的病理学意义。在神经退行性疾病进程中,细胞内溶酶体功能障碍导致的局部酸化,叠加神经炎症引起的温度升高,可能共同创造了一个触发蛋白质错误折叠的危险环境。RapiBOX平台所揭示的这种双重压力协同机制,为理解帕金森病的分子起源提供了新的视角。
展望:迈向单细胞蛋白质动态分析
这项工作代表了蛋白结构质谱分析技术的重要突破。传统方法如X射线晶体学、核磁共振和冷冻电镜虽然能够提供精细的结构信息,但通常需要高度纯化的样品且难以追踪动态过程。RapiBOX平台填补了这一空白,实现了在接近生理条件下观察蛋白质动态行为的能力。
李功玉教授表示,该平台的下一个目标是将分析能力推进到单个活细胞水平。这将使研究者能够超越群体平均效应,直接观察细胞个体之间的蛋白质行为异质性——这对于理解疾病发生机制和开发精准治疗策略具有重要价值。
南开大学化学学院2022级博士研究生刘军为该文第一作者,李功玉研究员为本文通讯作者,南开大学有机新物质创造前沿科学中心为论文通讯单位。中国科学技术大学刘扬中教授、安徽农业大学曹开明教授、天津医科大学郑珍教授和南开大学王倩倩、刘宁老师,以及Waters公司许译升工程师对本项目提供了重要的技术支持。本文主要质谱数据均采集于《天津物质绿色创造与制造海河实验室》结构质谱分析平台。本研究工作获国家重点研发计划青年科学家项目资助。
论文信息
Time‐Resolved In‐Cell Protein Interactions and Structural Dynamics via Rapid Buffer Online Exchange-Ion Mobility-Mass Spectrometry. Jun Liu, Xiaoli Wang, Xiangjun Si, Rui Zhao, Weida Qin, and Gongyu Li*. Angew. Chem. Int. Ed. 2025, e18076. https://doi.org/10.1002/anie.202518076




